
科学家利用基因编辑转座子改良水稻性状
转座子(TEs)是真核生物基因组中广泛存在的DNA重复序列,约占水稻基因组的35%。转座子是植物产生遗传变异的重要来源,通过多种机制调控基因表达及表型变异。水稻的泛转座子变异图谱研究表明,转座子在水稻驯化和育种性状改良方面发挥重要作用。
近日,中国科学院院士、遗传与发育生物学研究所研究员李家洋带领的科研团队,联合崖州湾国家实验室的研究人员,在《植物生物技术杂志》(Plant Biotechnology Journal)上在线发表了题为Generation of OsGRF4 and OsSNAC1 alleles for improving rice agronomic traits by CRISPR/Cas9-mediated manipulation of transposable elements的研究论文。该研究通过对水稻基因OsGRF4或OsSNAC1的非编码区进行转座子编辑,实现了对目的基因表达的精确调控。同时,该研究创制的优良等位基因为作物遗传育种提供了新策略。
微型反向重复转座子(MITEs)是短小而非自主的DNA转座子,是水稻基因组中数量较多的转座元件,且与至少58%的水稻基因相关。研究表明,MITEs是水稻基因表达变异的主要驱动因素之一,而利用MITE插入多态性进行全基因组关联研究有助于挖掘并控制农艺性状的潜在基因。
该研究推测,通过CRISPR/Cas9基因编辑技术设计基因非编码区的MITEs转座子分布可以上调或下调目标基因的表达,从而创制新的基因等位基因型以改良水稻性状。为验证这一设想,科研人员选择水稻中的生长调节因子4基因OsGRF4和胁迫响应基因OsSNAC1进行研究。研究显示,OsGRF4可正向调控水稻产量的相关性状,在其终止密码子下游的1200bp内插入一个294-bp的PIF/Harbinger超家族MITE;OsSNAC1可以增强水稻的耐盐性,但在其上下游非翻译区未检测到MITE。研究发现,水稻某些基因下游非编码区中的MITE可以介导靶基因的翻译抑制。因此,研究认为,通过CRISPR/Cas9技术删除OsGRF4下游非翻译区中的MITE,可以创制出过表达的等位基因型。研究针对OsGRF4基因的MITE靶区域,设计构建了2个CRISPR/Cas9 sgRNAs,并对其进行编辑。科研人员对得到的无转基因的纯合突变体进行分析发现,OsGRF4基因的MITE删除,提高了OsGRF4mite突变体中靶蛋白的丰度,并改善了与产量相关的农艺现状。水稻基因上游非翻译区中的一些MITEs可作为增强子,如miniature Ping (mPing) TE可以增强盐胁迫响应基因的转录水平。因此,研究人员尝试将430-bp的mPing插入耐盐基因OsSNAC1的上游非翻译区。进而,科研人员剖析得到的纯合突变体发现,OsSNAC1基因的MITE插入,提升了盐胁迫下OsSNAC1MITE突变体中靶基因的转录水平,并增强了它的耐盐性。
上述成果为转座子驱动的作物遗传育种提供了新途径。
研究工作得到科技创新2030-重大项目、国家自然科学基金及海南省相关项目的支持。
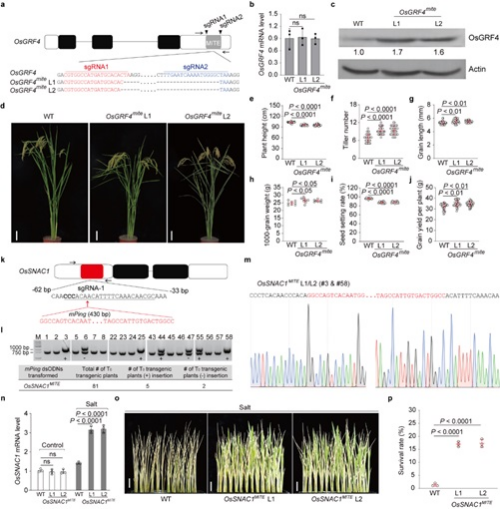
通过基因编辑技术设计非编码区的MITEs转座子分布来改良水稻性状
上一条:古蝉形态空间和空气动力学分析揭示中生代“飞行竞赛” 下一条:心理所发现人类嗅觉时间敏感性
【关闭】

